
RNA引导的CRISPR/Cas9系统是最简单、高效的基因编辑工具,但是它能够编辑的位点需要含有一个叫做PAM的序列,限制了其编辑范围。常用的SpCas9识别NGG PAM,经过多轮改造后几乎可以识别任何PAM。但是用于基因治疗时,SpCas9太大无法用单个AAV递送。随后,科学家又开发了较小的SaCas9。SaCas9识别的PAM是NNGRRT,编辑范围很小,无法广泛使用。还有一些Cas9被陆续开发出来,但是总体活性不高。另外,先前的Cas9工具都是外国科学家开发的,国内在这方面还是空白。
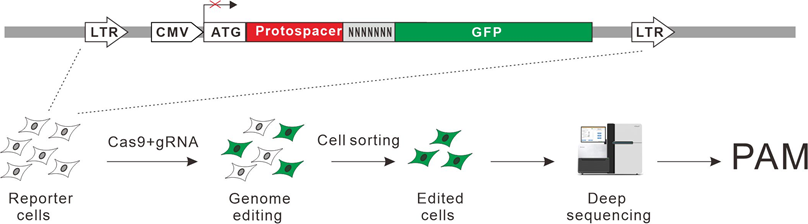
我室王永明课题组致力于开发小型Cas9系统,扩大编辑范围,填补国内空白。公共数据库中有上千种Cas9序列,但是大多数在人类细胞中都不工作。鉴定工作耗时耗力,成为开发工具的瓶颈。王永明课题组提出了鉴定Cas9活性的新思路,将基因编辑和GFP表达偶联,可以快速判定一个Cas9是否工作。
|
|
|
图1. Cas9活性鉴定思路 |
先后对300多个Cas9进行鉴定,开发出一系列性能优异的工具酶。2020年3月开发出国内首个SauriCas9,大小和活性与SaCas9相当,识别NNGG PAM,扩大了编辑范围。SauriCas9不足之处是特异性中等。2021年4月该课题组又开发出SlugCas9-HF,除了具备SauriCas9的优点,特异性还得到了极大的提高。SlugCas9-HF也是目前唯一一个活性可以和SpCas9姘美的工具酶。2021年12月该课题组又开发出SchCas9,识别更简单的NNGR(R=A or G) PAM,编辑范围在原来的基础上又翻了一倍。值得注意的是,上述工具酶都属于Type II-A Cas9,PAM偏好嘌呤碱基。
2022年8月12日,课题组又有新突破,在eLife上在线发表了题为“Closely related type II-C Cas9 orthologs recognize diverse PAMs”的研究论文。研究鉴定了25个Cas9的PAM,首次揭示了蛋白同源度很高的Type II-C Cas9识别PAM的多样性。这些PAM长度在5到11个碱基之间,包括富含C、T和A的PAM,变化比Type II-A Cas9更为丰富。论文还探讨了PAM识别和蛋白序列之间的规律。这些结果有助于开发新的Cas9工具。
|
|
|
图2. PAM多样性 |
随后,研究团队深入研究了Nsp2Cas9,它识别N4CC PAM,编辑活性和Nme2Cas9相当。
|
|
|
图3. Nsp2Cas9的PAM及编辑活性 |
在此基础上,作者又开发了Nsp2-SmuCas9嵌合体,识别N4C PAM,是PAM最简单的Cas9(图4)。Nsp2-SmuCas9含有1072个氨基酸,可以被AAV递送,对基因治疗具有重要的应用前景。总而言之,该研究揭示了Type II-C Cas9 PAM的多样性,为开发CRISPR-Cas9工具、扩大编辑范围提奠定了基础。
|
|
|
图4. Nsp2-SmuCas9构建示意图及其在内源位点的编辑效率 |
博士生魏京京是论文第一作者,王永明教授和复旦大学附属儿科医院孙淑娜副主任医师是论文的共同通讯作者。